最近一直在测试大模型来做生物信息,效果还可以,主要使用gemini cli,由于一直还有机会用上claude code,所以只能通过cursor来使用claude 4,这次内容我们来测试一下claud4的生物信息能力。
如何选择大模型
当前的大语言模型可谓是“百家争鸣,百花齐放”。各家都开足马力不断升级。目前有免费的,也有很多收费的。下面列出当前时间点一些主流大模型。主要就分中国和美国。
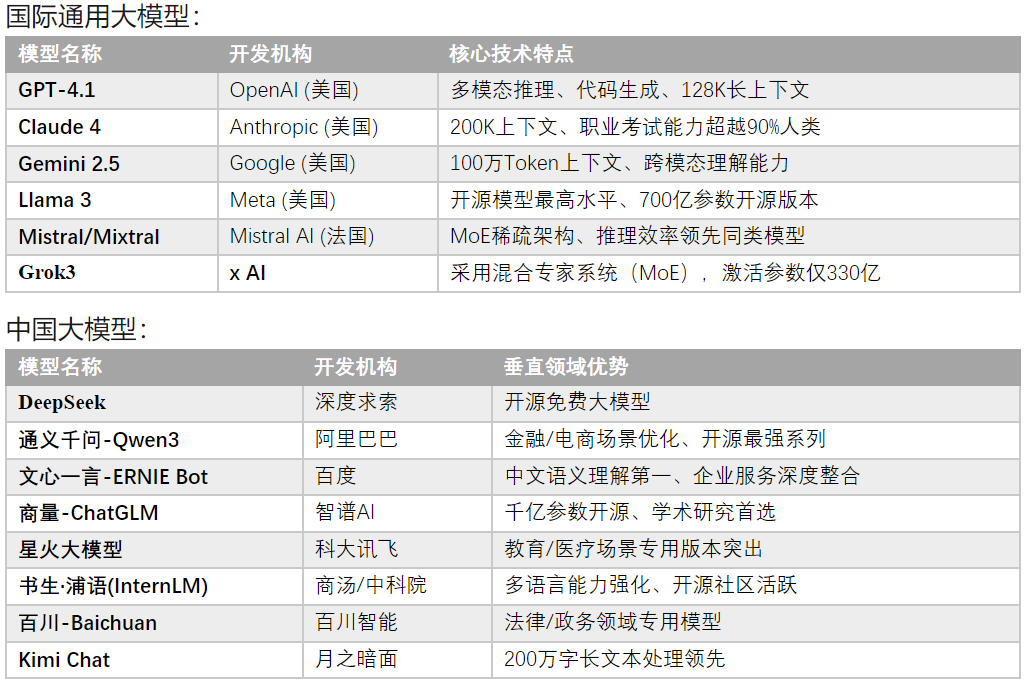
目前最强大的模型是Claude 4,但是需要付费,每月20美元左右。然后是gemini 2.5,有一定使用量限制,一般也够用,然后是完全免费的DeepSeek和Qwen3。
如何使用大模型
目前可以使用大模型的工具有很多,可以说非常混杂。
1、通过官网:这种模式只有聊天模式,生成代码需要手动复制粘贴,不方便生成测试和调试,不推荐。
2、插件模式:一般通过vscode或者jetbrain工具插件提供,插件提供了大量模型可供选择,例如trae ai,cline,CodeBuddy等,有免费有订阅。这种模式推荐vscode默认集成的copilot,每月订阅10美元,普通用户每月有一定量免费额度。
3、IDE模式:一般都是基于vscode开源版本开发,和vscode使用差别不大,例如cursor, windsurf,Trae都是这种模式,如果熟悉vscode,直接上手就可以,集成了agent功能,可以直接运行代码,不过需要订阅,每月10美元-20美元。
4、命令行模式:最新推出的Claude code,gemini cli都属于命令行模式,虽然很多人不喜欢命令行模式,但做生物信息这是最好的模式了。本来做分析也需要命令行,直接集成AI使用体验最好。
测试cluade 4生物信息分析能力
由于claude 4对国内IP封禁,因此我订阅了cursor,通过cursor来测试claude 4模型。
我们通过cursor的ssh功能登录服务器,就可以直接调用agent或者命令行直接运行代码。
这里我们想使用kraken2对宏基因组测测序数据mocklog.fastq.gz进行物种分类,解读报告,数据可视化以及提取序列。
首先,输入提示词“对纳米孔测序/ifs1/User/cursor/02.kraken/mocklog.fastq.gz进行物种分类”
选择claude sonnet4模型,cursor接到任务之后,先确认文件,然后确认分析软件和数据库,都存在之后直接生成kraken2分析命令。选择kraken2以及对应的脚本都是有模型默认生成的。
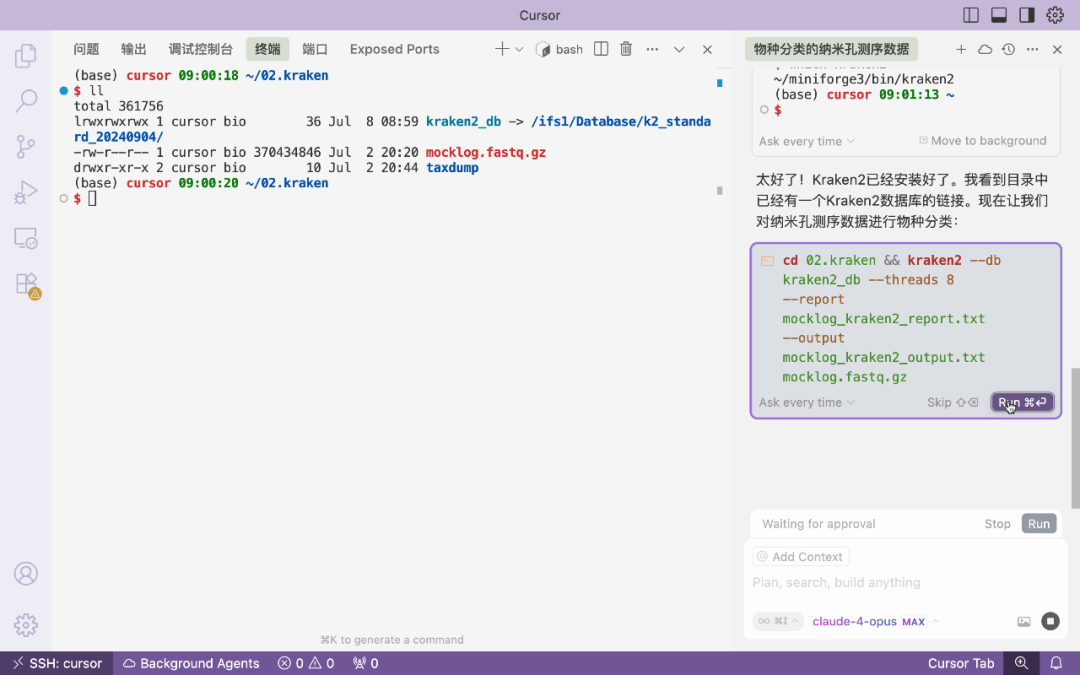
分析完成之后直接输入解读报告命令。cursor会对生成结果进行中文解读,非常方便。
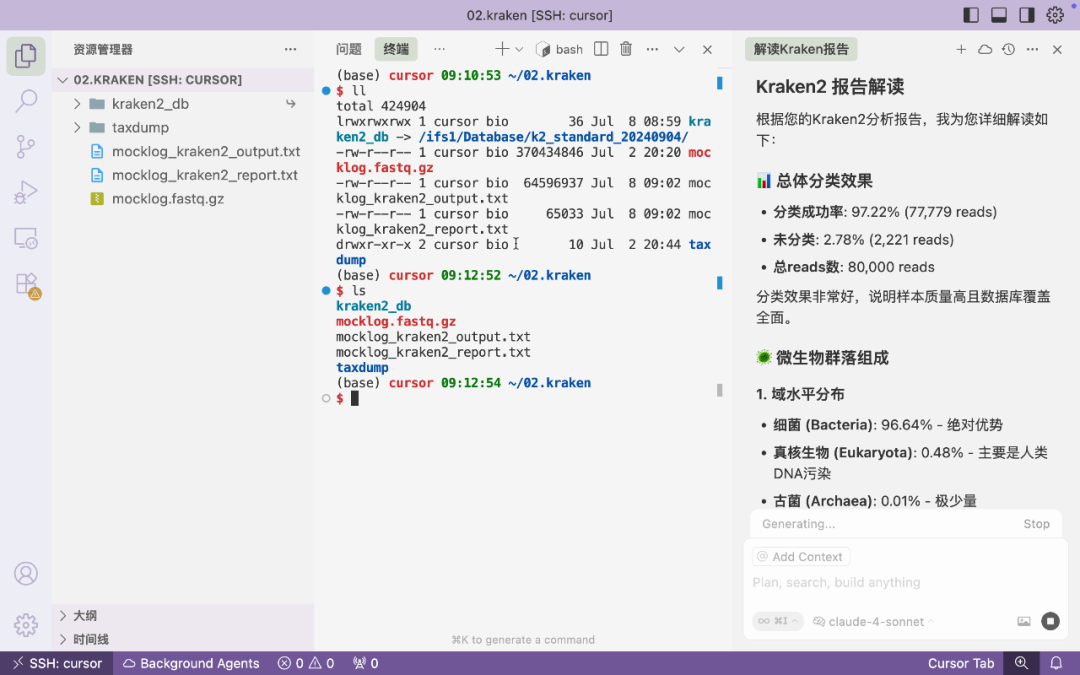
接下来要求数据可视化。
cursor直接生成一个python程序用来做数据分析,由于缺少一些依赖包,pyhton选择自己安装。
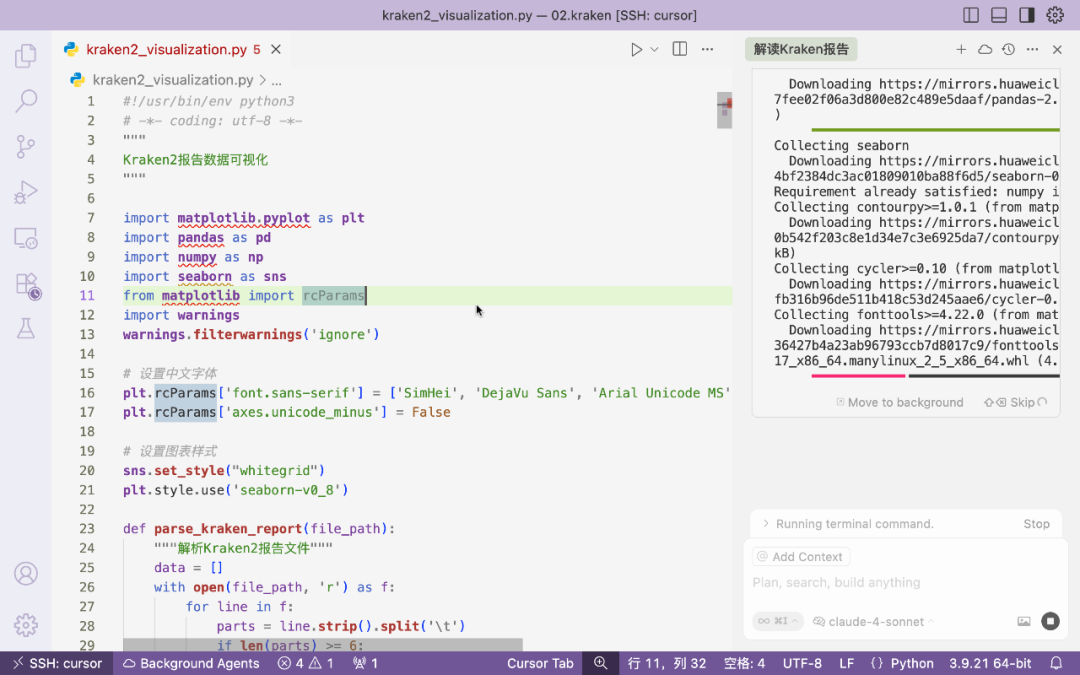
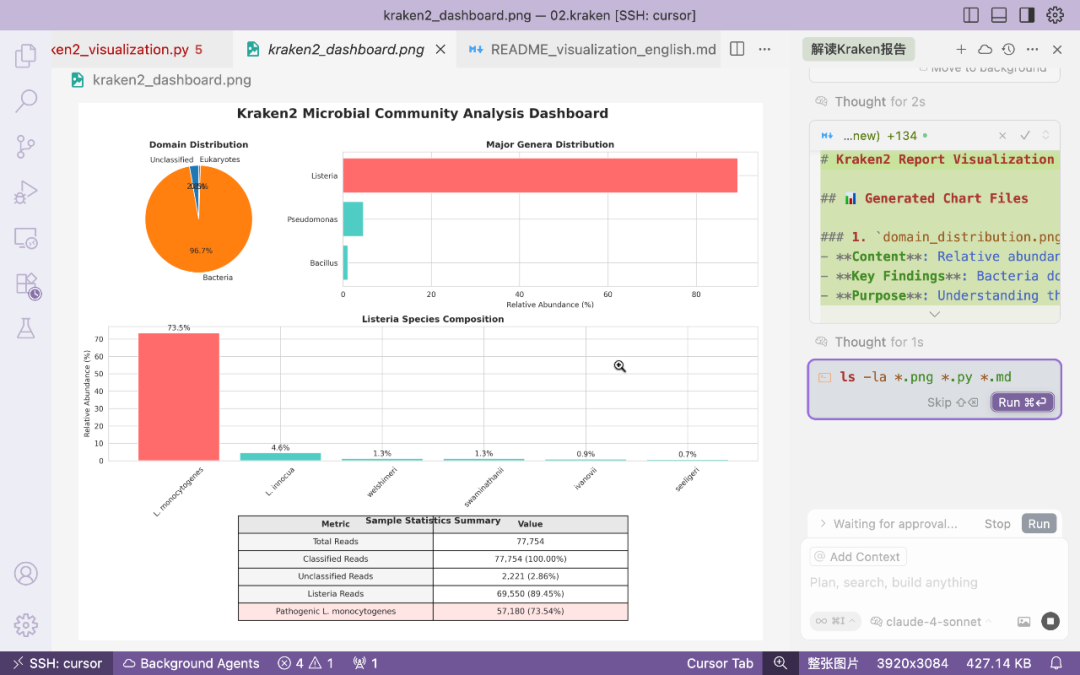
配置结束后就开始绘图。直接生成了绘图结果以及一个markdown报告。
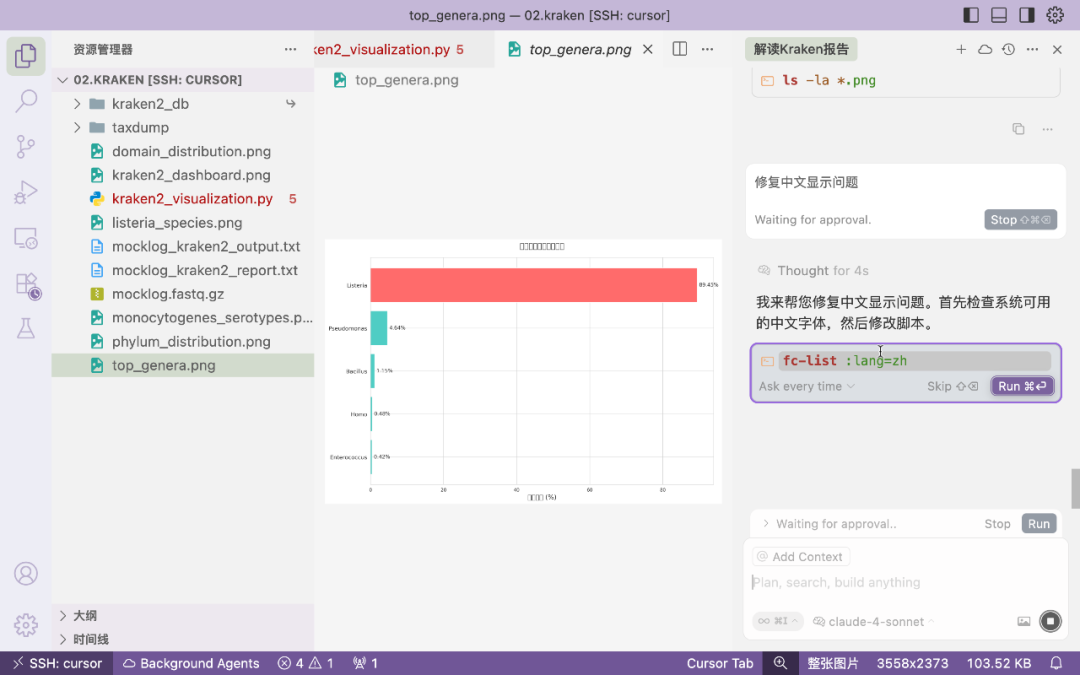
不过图片标签中无法显示中文,让cursor自己修复,修复完还是有问题,于是直接让它将标签都换成英文,这次显示没有问题了。
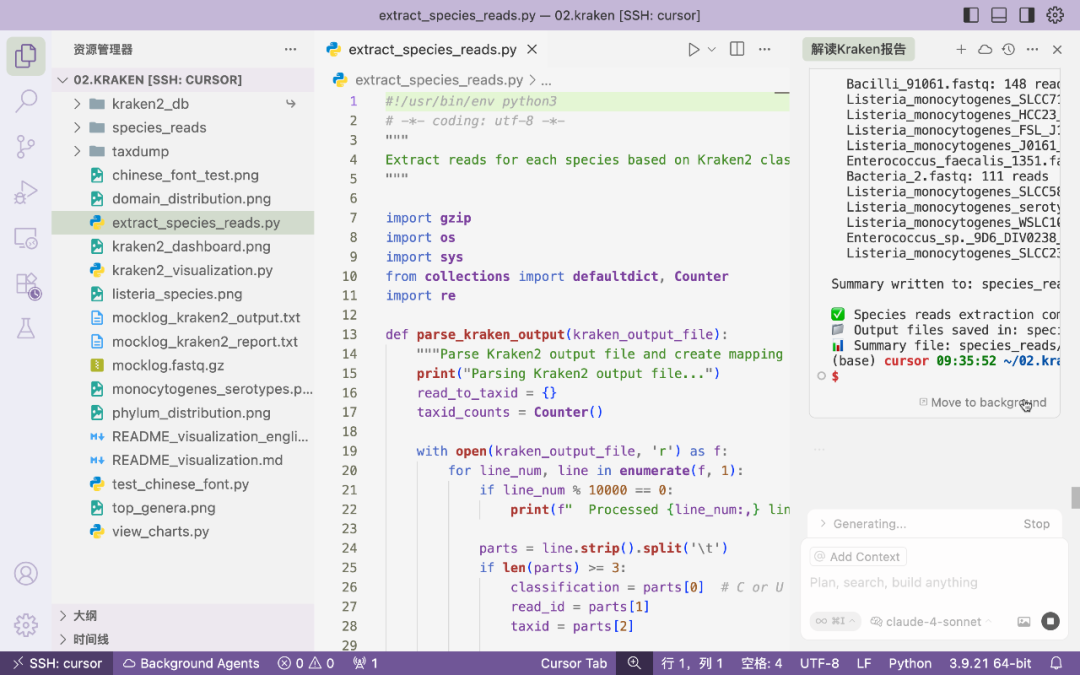
最后我们提取某个每个物种的比对序列。cursor又是自己写个程序完成工作。
cursor总结
通过curor来远程访问服务器,然后调用模型来做生物信息要比直接通过命令行方便很多,很多新手还是习惯这种有图形化的操作。切换模型也很方便,比直接用gemini cli方便一些,唯一不足的地方是cursor并不免费。不过cursor调用这些大模型都是要收费的,这点完全可以理解,免费的往往是最贵的。
这些工具确实非常好用,但是并不能让零基础人员正确使用,也就是它是完成60-90分的过程,而并不是从0到60的过程。
文章来自于微信公众号“基因学苑”。

